BIOPACで測定したECGデータをKubiosで解析する
ここでは、Biopacを用いてECGを計測しKubiosを用いて、各種の心拍変動解析を実施する方法を説明します。各手順の詳しい方法は、動画の下の解説をご覧ください。
BIOPACでECGを測定
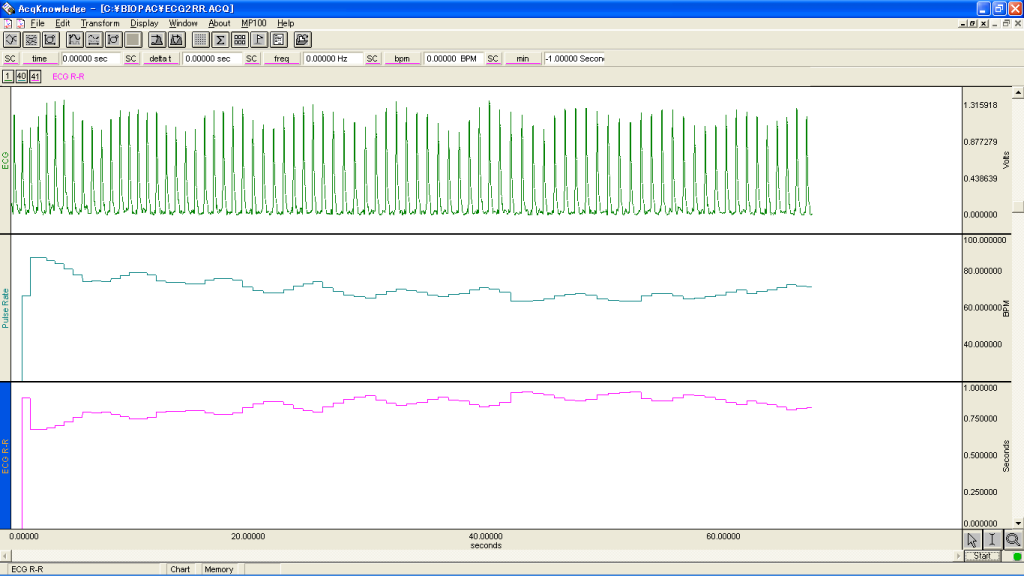
BiopacのMP100を用いて、1チャンネル目にECG、2チャンネル目にHR、3チャンネル目にRR間隔を表示・計測します。
データの出力と形式の確認
「File>Save as」でデータを保存すると、ECG、HR、RR間隔の各値が、200Hzのサンプル周波数で、タブ区切りで保存されていることがわかります。以下のPythonスクリプトを用いて、ECG波形からRR間隔を算出してみましょう。
#ECG2RR.py
#Transfer ECG wave data to RR interval data
import sys
import os
import numpy as np
from scipy.signal import find_peaks
import matplotlib.pyplot as plt
# コマンドライン引数からファイル名を取得
file_name = sys.argv[1]
# ファイルの読み込み
data = np.genfromtxt(file_name, delimiter='\t', usecols=[0, 1, 2])
# 1列目のデータの取り出し
ecg = data[:, 0]
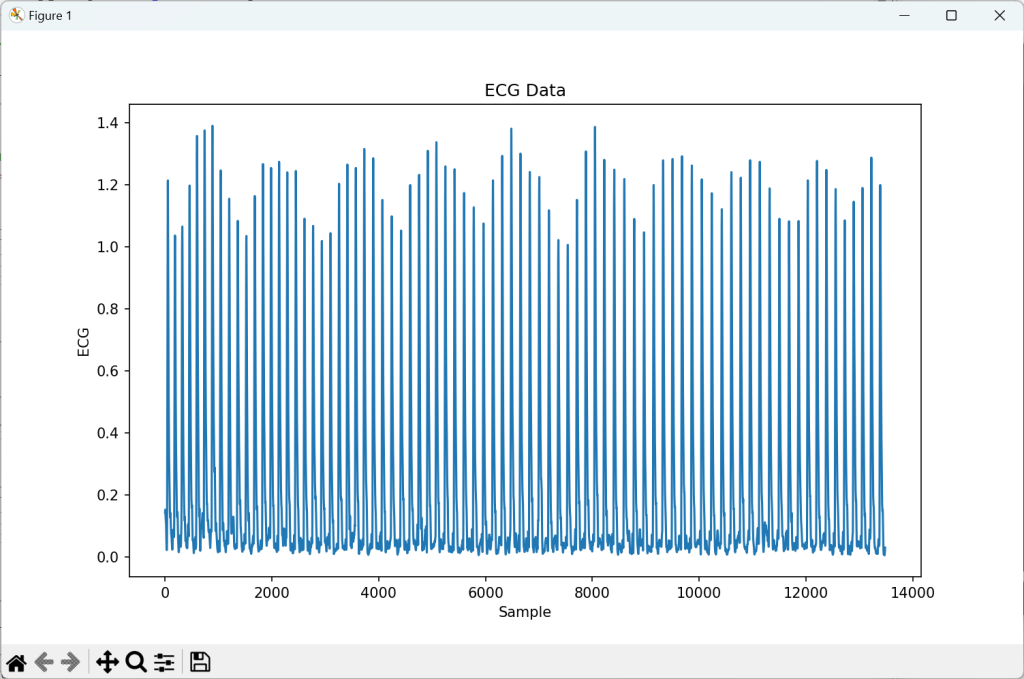
plt.figure(figsize=(10, 6)) # 幅: 10インチ, 高さ: 6インチ
plt.plot(ecg)
plt.xlabel('Sample')
plt.ylabel('ECG')
plt.title('ECG Data')
plt.show(block=True)
# ピークの検出
peaks, _ = find_peaks(ecg, height=0.5)
# ピーク間隔の計算
peak_intervals = np.diff(peaks) * 0.005 * 1000 # convert to milliseconds
#print(peak_intervals)
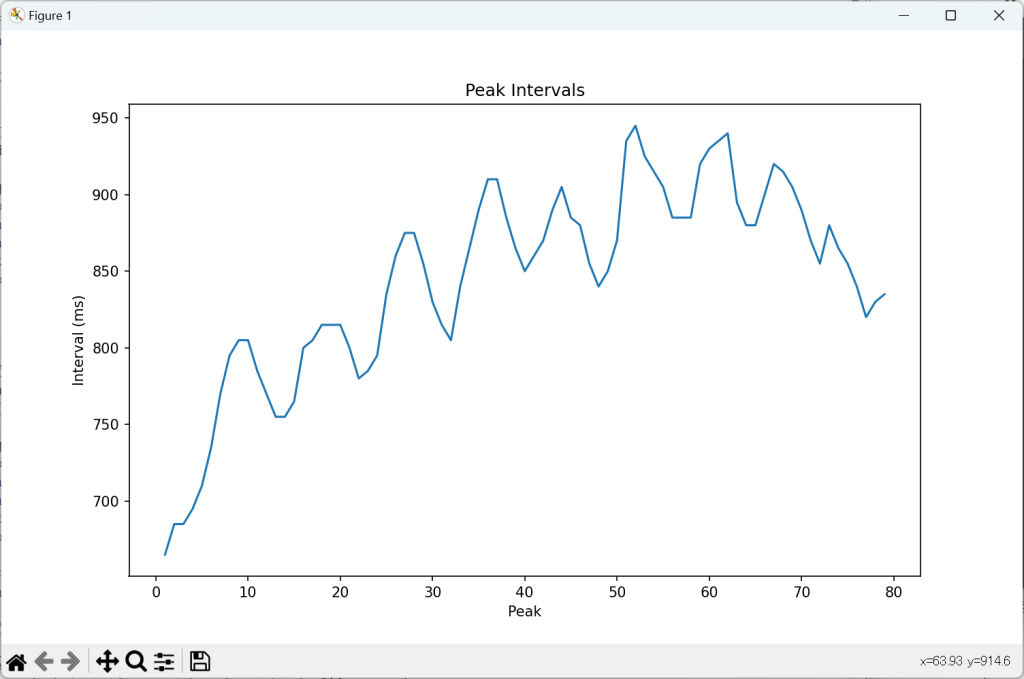
plt.figure(figsize=(10, 6), dpi=100) # 幅: 10インチ, 高さ: 6インチ, dpi: 100
plt.plot(np.arange(1, len(peaks)), peak_intervals)
plt.xlabel('Peak')
plt.ylabel('Interval (ms)')
plt.title('Peak Intervals')
plt.show(block=True)
# ファイル名の拡張子を除いた部分を取得
file_name_base = os.path.splitext(file_name)[0]
# ピーク間隔を保存
output_file_name = file_name_base + '_RR.csv'
np.savetxt(output_file_name, peak_intervals, delimiter=',', fmt='%.2f')
print('Peak intervals saved to', output_file_name)
このスクリプトを使って「ECGdata1.txt」というファイルを処理する際は、「python ECG2RR.py ECGdata1.txt」のようにコマンドを打ち込みます。すると、データが読み込まれ、ECG波形が表示された後、検出されたRR間隔データが表示されます。RR間隔データは、「ECGdata1_RR.csv」という名称で同一階層に保存されます。
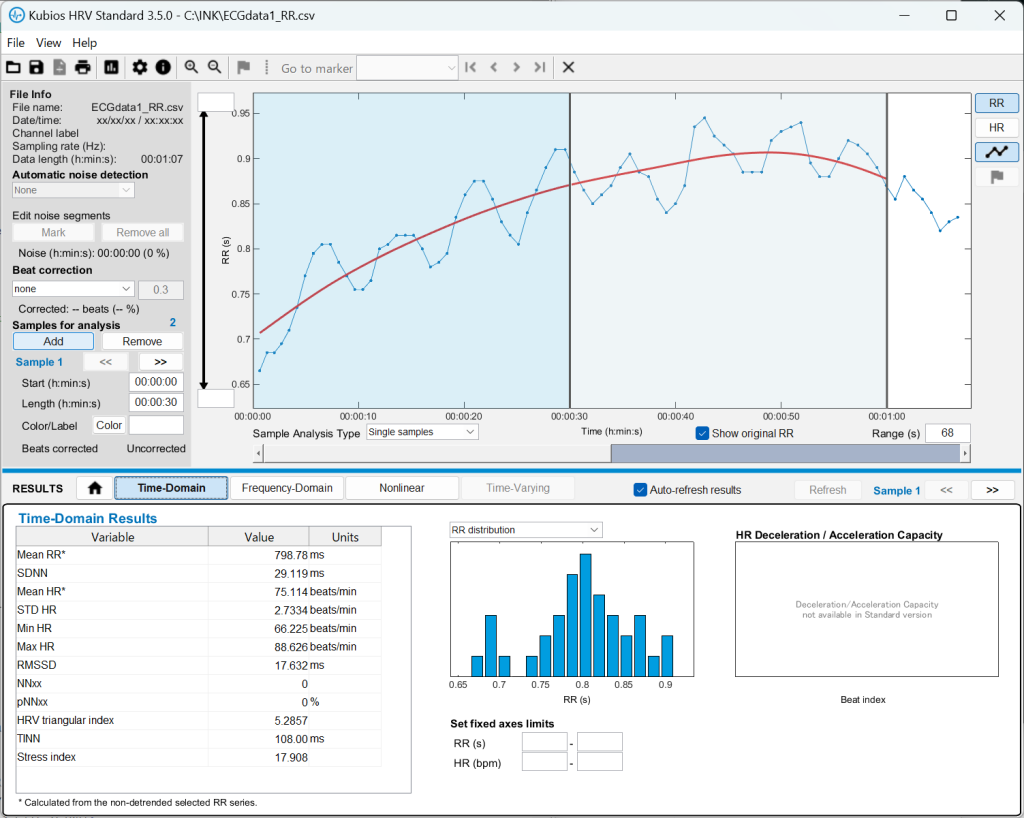
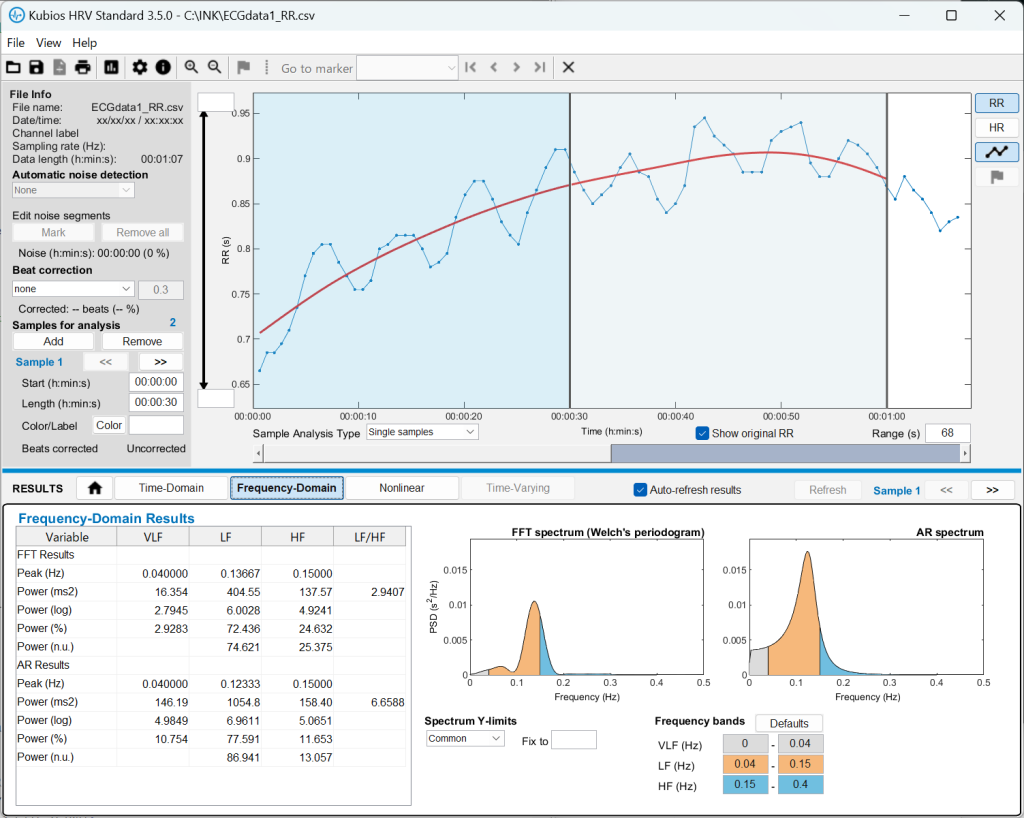
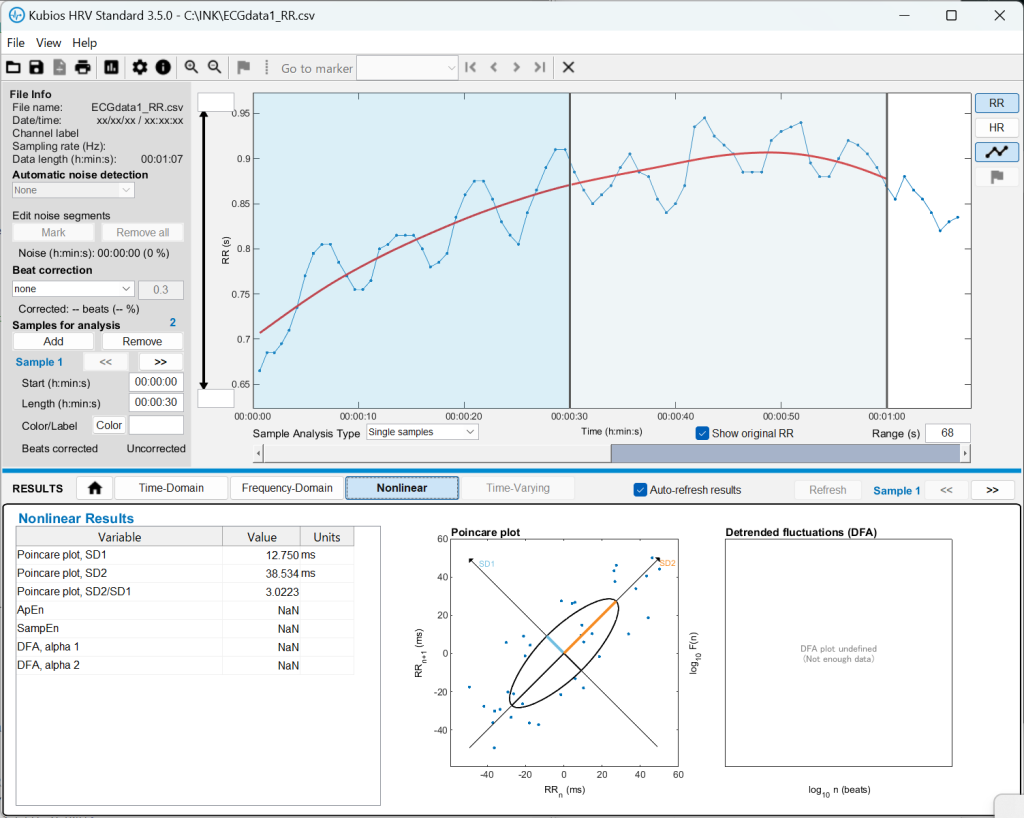
Kubiosを用いた心拍変動解析
上記で出力されたRR間隔データをKubiosで解析してみましょう。解析を行う範囲を指定すると、画面下方に「Time-domain」、「Frequency-domain」、「Nonlinear」の解析結果が表示されます。解析範囲を切り替えながら、各評価地をチェックしていきましょう。